Eine neue Studie enthüllt einen Mechanismus zur Erzeugung von DNA-Palindromen, der zu neuen microRNA-Genen führen könnte, Licht auf die Herkunft der Gene wirft und möglicherweise unser Verständnis von RNA-Strukturen beeinflusst.
Forscher der Universität Helsinki haben einen Mechanismus entdeckt, der eine sofortige Generierung bewirkt DNA Palindrome, die möglicherweise zur Entstehung neuer microRNA-Gene aus nichtkodierenden DNA-Sequenzen führen. Diese Entdeckung wurde bei der Untersuchung von DNA-Replikationsfehlern und deren Auswirkungen gemacht RNA Molekülstrukturen bietet neue Einblicke in die Genesursprünge.
Die Komplexität lebender Organismen ist in ihren Genen verankert, aber woher kommen diese Gene? Forscher der Universität Helsinki haben offene Fragen zum Ursprung kleiner regulatorischer Gene gelöst und einen Mechanismus beschrieben, der ihre DNA-Palindrome erzeugt. Unter geeigneten Umständen entwickeln sich diese Palindrome zu microRNA-Genen.
Gene und Proteine: Die Bausteine des Lebens
Das menschliche Genom enthält ca. 20.000 Gene, die für den Aufbau von Proteinen verwendet werden. Die Aktionen dieser klassischen Gene werden von Tausenden regulatorischen Genen koordiniert, von denen die kleinsten microRNA-Moleküle mit einer Länge von 22 Basenpaaren kodieren. Während die Anzahl der Gene relativ konstant bleibt, entstehen im Laufe der Evolution gelegentlich neue Gene. Ähnlich wie die Entstehung des biologischen Lebens fasziniert die Entstehung neuer Gene die Wissenschaftler nach wie vor.
Das palindromische Rätsel lösen
Alle RNA-Moleküle benötigen palindromische Basenreihen, die das Molekül in seiner funktionellen Konformation fixieren. Wichtig ist, dass die Wahrscheinlichkeit, dass zufällige Basenmutationen nach und nach solche palindromischen Läufe bilden, äußerst gering ist, selbst bei den einfachen microRNA-Genen. Daher gibt der Ursprung dieser palindromischen Sequenzen den Forschern Rätsel auf. Experten am Institut für Biotechnologie der Universität Helsinki, Finnland, haben dieses Rätsel gelöst und einen Mechanismus beschrieben, der sofort vollständige DNA-Palindrome erzeugen und so neue microRNA-Gene aus zuvor nicht kodierenden DNA-Sequenzen erzeugen kann.
Einblicke in die DNA-Replikation
In einem von der Akademie von Finnland finanzierten Projekt untersuchten Forscher Fehler bei der DNA-Replikation. Ari Löytynoja, der Projektleiter, vergleicht die DNA-Replikation mit dem Tippen von Text.
„Die DNA wird eine Base nach der anderen kopiert, und typischerweise handelt es sich bei Mutationen um fehlerhafte einzelne Basen, wie Fehleingaben auf einer Laptop-Tastatur. Wir haben einen Mechanismus untersucht, der größere Fehler verursacht, etwa das Kopieren und Einfügen von Text aus einem anderen Kontext. Besonders interessierten uns Fälle, in denen der Text rückwärts kopiert wurde, sodass ein Palindrom entstand.“
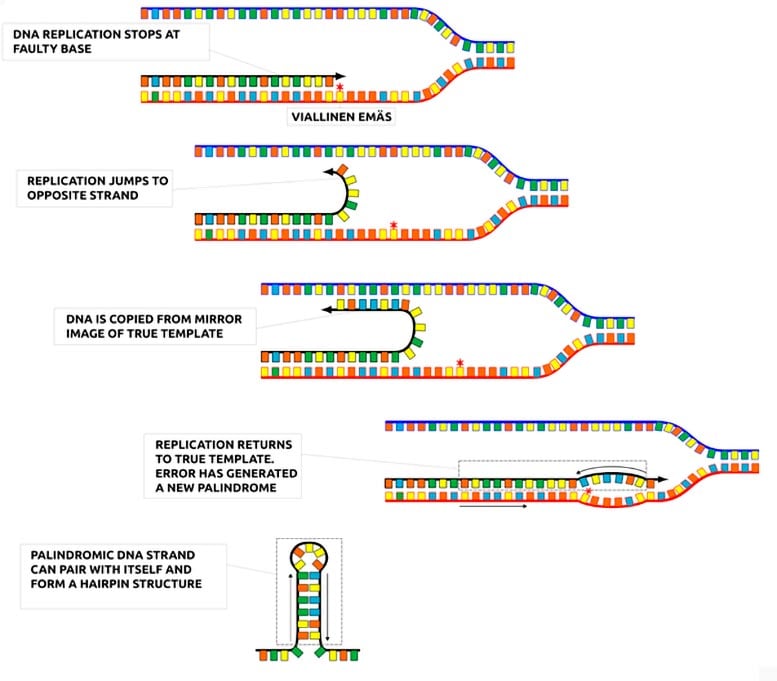
Forscher untersuchten einen Fehlermechanismus bei der DNA-Replikation und stellten fest, dass einige Fehler Palindrome erzeugen, die sich zu Haarnadelstrukturen falten können. Bildnachweis: Ari Löytynoja
RNA-Strukturen und DNA-Fehler
Forscher erkannten, dass DNA-Replikationsfehler manchmal von Vorteil sein könnten. Sie berichteten Mikko Frilander, einem Experten für RNA-Biologie, über diese Ergebnisse. Er erkannte sofort den Zusammenhang mit der Struktur von RNA-Molekülen.
„In einem RNA-Molekül können sich die Basen benachbarter Palindrome paaren und Strukturen bilden, die einer Haarnadel ähneln. Solche Strukturen sind entscheidend für die Funktion der RNA-Moleküle“, erklärt er.
Aufgrund ihrer einfachen Struktur entschieden sich die Forscher, sich auf microRNA-Gene zu konzentrieren: Die Gene sind sehr kurz – nur wenige Dutzend Basen – und müssen sich zu einer Haarnadelstruktur falten, um richtig zu funktionieren.
Eine zentrale Erkenntnis bestand darin, die Gengeschichte mithilfe eines benutzerdefinierten Computeralgorithmus zu modellieren. Laut Postdoktorand Heli Mönttinen ermöglicht dies die bislang genaueste Untersuchung der Herkunft von Genen.
„Das gesamte Genom von Dutzenden Primaten und Säugetieren ist bekannt. Ein Vergleich ihrer Genome verrät, welche Spezies das microRNA-Palindrompaar haben und denen es fehlt. Mit einer detaillierten Modellierung der Geschichte könnten wir sehen, dass durch einzelne Mutationsereignisse ganze Palindrome entstehen“, sagt Mönttinen.
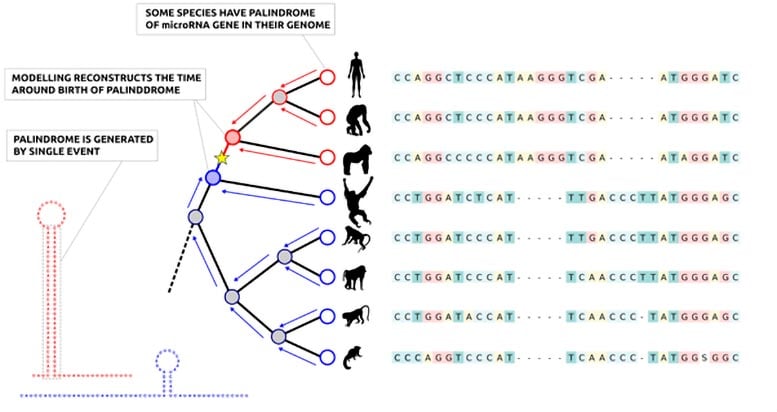
Eine zentrale Erkenntnis bestand darin, die Gengeschichte anhand von Informationen verwandter Arten zu modellieren. Die Modellierung zeigte, dass die Palindrome von microRNA-Genen durch einzelne Mutationsereignisse erzeugt werden. Bildnachweis: Ari Löytynoja
Implikationen und Universalität
Indem sie sich auf Menschen und andere Primaten konzentrierten, zeigten Forscher in Helsinki, dass der neu entdeckte Mechanismus mindestens ein Viertel der neuen microRNA-Gene erklären kann. Da ähnliche Fälle in anderen Evolutionslinien gefunden wurden, scheint der Ursprungsmechanismus universell zu sein.
Im Prinzip ist der Aufstieg von microRNA-Genen so einfach, dass neue Gene Auswirkungen auf die menschliche Gesundheit haben könnten. Heli Mönttinen sieht die Bedeutung der Arbeit weiter gefasst, beispielsweise im Verständnis der Grundprinzipien des biologischen Lebens.
„Die Entstehung neuer Gene aus dem Nichts hat Forscher fasziniert. Wir haben jetzt ein elegantes Modell für die Evolution von RNA-Genen“, betont sie.
Obwohl die Ergebnisse auf kleinen regulatorischen Genen basieren, glauben Forscher, dass die Ergebnisse auf andere RNA-Gene und -Moleküle übertragen werden können. Durch die Verwendung der durch den neu entdeckten Mechanismus erzeugten Rohstoffe kann die natürliche Selektion beispielsweise viel komplexere RNA-Strukturen und -Funktionen schaffen.
Die Studie wurde veröffentlicht in PNAS.
Referenz: „Generierung von De-novo-miRNAs durch Template-Wechsel während der DNA-Replikation“ von Heli AM Mönttinen, Mikko J. Frilander und Ari Löytynoja, 29. November 2023, Verfahren der Nationalen Akademie der Wissenschaften.
DOI: 10.1073/pnas.2310752120

