Forscher der UC San Diego stellen Multi-Scale Integrated Cell (MuSIC) vor, eine Technik, die Mikroskopie, Biochemie und künstliche Intelligenz kombiniert und bisher unbekannte Zellkomponenten aufdeckt, die neue Hinweise auf menschliche Entwicklung und Krankheiten geben können. (Konzeptuelle Darstellung des Künstlers.) Bildnachweis: UC San Diego Health Sciences
Eine auf künstlicher Intelligenz basierende Technik enthüllt bisher unbekannte Zellbestandteile, die neue Hinweise auf die menschliche Entwicklung und Krankheit liefern könnten.
Die meisten menschlichen Krankheiten können auf fehlerhafte Teile einer Zelle zurückgeführt werden – ein Tumor kann wachsen, weil ein Gen nicht genau in ein bestimmtes Protein übersetzt wurde, oder eine Stoffwechselerkrankung entsteht, weil Mitochondrien nicht richtig feuern. Um jedoch zu verstehen, welche Teile einer Zelle bei einer Krankheit schief gehen können, benötigen Wissenschaftler zunächst eine vollständige Liste der Teile.
Durch die Kombination von Mikroskopie, biochemischen Techniken und künstlicher Intelligenz haben Forscher der University of California San Diego School of Medicine und ihre Mitarbeiter einen bedeutenden Fortschritt beim Verständnis menschlicher Zellen gemacht.
Die als Multi-Scale Integrated Cell (MuSIC) bekannte Technik wird am 24. November 2021 in . beschrieben Natur.
„Wenn Sie sich eine Zelle vorstellen, stellen Sie sich wahrscheinlich das bunte Diagramm in Ihrem Zellbiologie-Lehrbuch vor, mit Mitochondrien, endoplasmatischem Retikulum und Zellkern. Aber ist das die ganze Geschichte? Definitiv nicht“, sagte Trey Ideker, PhD, Professor an der UC San Diego School of Medicine und am Moores Cancer Center. “Wissenschaftler haben schon lange erkannt, dass wir mehr nicht wissen als wir wissen, aber jetzt haben wir endlich einen Weg, tiefer zu schauen.”
Ideker leitete die Studie zusammen mit Emma Lundberg, PhD, vom KTH Royal Institute of Technology in Stockholm, Schweden, und der Stanford University.
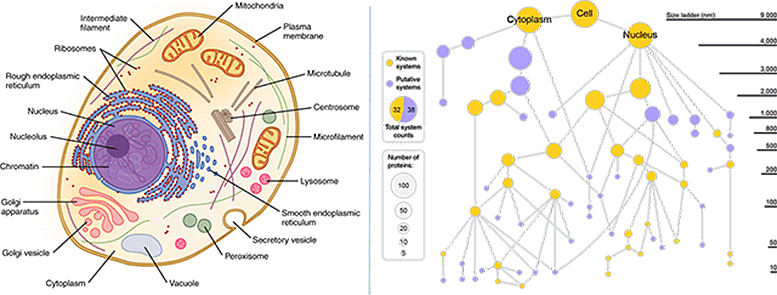
Links: Klassische Zelldiagramme aus Lehrbüchern implizieren, dass alle Teile klar sichtbar und definiert sind. (Quelle: OpenStax/Wikimedia). Rechts: Eine mit MuSIC-Technik generierte neue Zellkarte zeigt viele neuartige Komponenten. Goldene Knoten repräsentieren bekannte Zellkomponenten, violette Knoten repräsentieren neue Komponenten. Die Größe des Knotens spiegelt die Anzahl der unterschiedlichen Proteine in dieser Komponente wider. Bildnachweis: UC San Diego Health Sciences
In der Pilotstudie zeigte MuSIC etwa 70 Komponenten, die in einer menschlichen Nierenzelllinie enthalten waren, von denen die Hälfte noch nie zuvor gesehen wurde. In einem Beispiel entdeckten die Forscher eine Gruppe von Proteinen, die eine unbekannte Struktur bilden. In Zusammenarbeit mit dem Kollegen Gene Yeo, PhD von der UC San Diego, stellten sie schließlich fest, dass die Struktur ein neuer Komplex von Proteinen ist, der bindet RNA. Der Komplex ist wahrscheinlich am Spleißen beteiligt, einem wichtigen zellulären Ereignis, das die Translation von Genen in Proteine ermöglicht und hilft zu bestimmen, welche Gene zu welchen Zeiten aktiviert werden.
Das Innere von Zellen – und die vielen dort gefundenen Proteine – werden typischerweise mit einer von zwei Techniken untersucht: Mikroskopische Bildgebung oder biophysikalische Assoziation. Bei der Bildgebung fügen Forscher fluoreszierende Markierungen in verschiedenen Farben zu interessierenden Proteinen hinzu und verfolgen ihre Bewegungen und Assoziationen über das Sichtfeld des Mikroskops. Um biophysikalische Assoziationen zu untersuchen, könnten Forscher einen spezifischen Antikörper für ein Protein verwenden, um es aus der Zelle zu ziehen und zu sehen, was sonst noch daran befestigt ist.
Das Team interessiert sich seit vielen Jahren dafür, das Innenleben von Zellen zu kartieren. Der Unterschied zu MuSIC ist die Verwendung von Deep Learning, um die Zelle direkt aus zellularen Mikroskopiebildern abzubilden.
„Die Kombination dieser Technologien ist einzigartig und leistungsstark, weil zum ersten Mal Messungen auf sehr unterschiedlichen Skalen zusammengeführt wurden“, sagte Yue Qin, Erstautor der Studie, ein Doktorand der Bioinformatik und Systembiologie in Idekers Labor.
Mikroskope ermöglichen es Wissenschaftlern, bis auf die Ebene eines einzelnen Mikrometers zu sehen, etwa so groß wie einige Organellen, wie zum Beispiel Mitochondrien. Kleinere Elemente, wie einzelne Proteine und Proteinkomplexe, können durch ein Mikroskop nicht gesehen werden. Biochemische Techniken, die mit einem einzigen Protein beginnen, ermöglichen es Wissenschaftlern, bis in den Nanometerbereich vorzudringen. (Ein Nanometer ist ein Milliardstel Meter oder 1.000 Mikrometer.)
„Aber wie überbrückt man diese Lücke vom Nanometer- bis zum Mikrometerbereich? Das ist seit langem eine große Hürde in den Biowissenschaften“, sagte Ideker, der auch Gründer der UC Cancer Cell Map Initiative und des UC San Diego Center for Computational Biology and Bioinformatics ist. „Es stellte sich heraus, dass Sie dies mit künstlicher Intelligenz tun können – indem Sie Daten aus mehreren Quellen betrachten und das System bitten, sie zu einem Modell einer Zelle zusammenzusetzen.“
Das Team trainierte die künstliche Intelligenzplattform MuSIC, um sich alle Daten anzusehen und ein Modell der Zelle zu erstellen. Das System ordnet die Zellinhalte noch nicht bestimmten Orten zu, wie z. B. einem Lehrbuchdiagramm, teilweise weil ihre Positionen nicht unbedingt fest sind. Stattdessen sind die Positionen der Komponenten fließend und ändern sich je nach Zelltyp und Situation.
Ideker stellte fest, dass dies eine Pilotstudie zum Testen von MuSIC war. Sie haben sich nur 661 Proteine und einen Zelltyp angesehen.
„Der klare nächste Schritt ist, die gesamte menschliche Zelle zu durchblasen“, sagte Ideker, „und dann zu verschiedenen Zelltypen, Menschen und Spezies überzugehen. Schließlich könnten wir die molekularen Grundlagen vieler Krankheiten besser verstehen, indem wir die Unterschiede zwischen gesunden und kranken Zellen vergleichen.“
Referenz: „A multi-scale map of cell structure fuusing protein images and interactions“ von Yue Qin, Edward L. Huttlin, Casper F. Winsnes, Maya L. Gosztyla, Ludivine Wacheul, Marcus R. Kelly, Steven M. Blue, Fan Zheng, Michael Chen, Leah V. Schaffer, Katherine Licon, Anna Bäckström, Laura Pontano Vaites, John J. Lee, Wei Ouyang, Sophie N. Liu, Tian Zhang, Erica Silva, Jisoo Park, Adriana Pitea, Jason F. Kreisberg, Steven P. Gygi, Jianzhu Ma, J. Wade Harper, Gene W. Yeo, Denis LJ Lafontaine, Emma Lundberg und Trey Ideker, 24. November 2021, Natur.
DOI: 10.1038/s41586-021-04115-9
Co-Autoren sind: Maya L. Gosztyla, Marcus R. Kelly, Steven M. Blue, Fan Zheng, Michael Chen, Leah V. Schaffer, Katherine Licon, John J. Lee, Sophie N. Liu, Erica Silva, Jisoo Park, Adriana Pitea, Jason F. Kreisberg, UC San Diego; Edward L. Huttlin, Laura Pontano Vaites, Tian Zhang, Steven P. Gygi, J. Wade Harper, Harvard Medical School; Casper F. Winsnes, Anna Bäckström, Wei Ouyang, KTH Royal Institute of Technology; Ludivine Wacheul, Denis LJ Lafontaine, Université Libre de Bruxelles; und Jianzhu Ma, Universität Peking.
Die Finanzierung dieser Forschung wurde teilweise von den National Institutes of Health (Zuschüsse U54CA209891, U01MH115747, F99CA264422, P41GM103504, R01HG009979, U24HG006673, U41HG009889, R01HL137223, R01HG004659, R50CA243885), Google Ventures, Family Erling-Pernuts Wallenberg Foundation (Grant 2016.0204), Swedish Research Council (Grant 2017-05327), Belgischer Fonds de la Recherche Scientifique, Université Libre de Bruxelles, European Joint Program on Rare Diseases, Région Wallonne, Internationale Brachet Stiftung und Epitran COST action (Grant CA16120 ).
Offenlegung: Trey Ideker ist Mitbegründer von, im wissenschaftlichen Beirat und hält eine Kapitalbeteiligung an Data4Cure, Inc. Ideker ist auch im wissenschaftlichen Beirat, hält eine Kapitalbeteiligung an Ideaya BioSciences, Inc. und erhält gesponserte Forschungsförderung von Ideaya BioSciences, Inc. Gene Yeo ist Mitbegründer, Mitglied des Board of Directors, im Scientific Advisory Board, Anteilseigner und bezahlter Berater von Locanabio und Eclipse BioInnovations. Yeo ist zudem Gastprofessor an der National University of Singapore. Die Bedingungen dieser Vereinbarungen wurden von der University of California San Diego in Übereinstimmung mit ihren Richtlinien zu Interessenkonflikten geprüft und genehmigt. Emma Lundberg ist Mitglied des wissenschaftlichen Beirats von Cartography Biosciences, Nautilus Biotechnology und Interline Therapeutics und hält Beteiligungen an diesen. J. Wade Harper ist Mitbegründer von, im wissenschaftlichen Beirat und hält eine Beteiligung an Caraway Therapeutics. Harper ist auch wissenschaftlicher Gründungsberater für Interline Therapeutics.

